IGV(Integrative Genomics Viewer)は、マッピングデータを視覚的に確認することができる大変便利なツールです。

IGVとは
IGVは、米Broad Instituteによって作成されたゲノムブラウザです。様々フォーマットのデータに対応し、直感的に操作できる使いやすいツールです。要Java動作環境。
IGV
IGVのインストールと起動
準備
IGV 2.3.xは、Java 7を要求します。今回は、Java 7/Java 8(Java 1.8へ更新)の両方で動作を確認しました。
$ java -version java version "1.7.0_80" Java(TM) SE Runtime Environment (build 1.7.0_80-b15) Java HotSpot(TM) 64-Bit Server VM (build 24.80-b11, mixed mode)
$ java -version java version "1.8.0_91" Java(TM) SE Runtime Environment (build 1.8.0_91-b14) Java HotSpot(TM) 64-Bit Server VM (build 25.91-b14, mixed mode)
インストール
今回は、ダウンロードのサイトから、「Binary Distribution」(IGV_2.3.80.zip)をダウンロードしました。zipファイルを展開してインストール完了です。
IGVを起動する
Linux(Ubuntu 14.04)で起動する際は、zipファイルを展開したフォルダに移動して、./igv.sh &で起動することが出来ます。
IGVの使い方
リファレンスとなるゲノムファイル(及び、インデックス付与したfaiファイル)と、マッピング結果のソート済みbamファイル(及び、インデックスを付与したbaiファイル)を準備します。ゲノムファイルについて、代表的な生物(Human, Mouse, Rat)に関しては、あらかじめIGVでプリセットデータがあります。しかし、マッピングで用いたゲノムファイルやアノテーション情報があれば、ファイルからロードすることも出来ます。

リファレンスファイルの読み込み
メニューバーのGenomes->Load Genome From File...から、fastaファイルをロードします。ロードが完了すると、メニューバー下の「switch the current genome」で、ロードしたゲノムへ切り替えできるようになります。(IGV:Loading a Genome)
IGVで表示しやすいように、samtoolsで事前にindexの付与を済まして置くと良いです。IGVでロードする際、genome.faを指定しますが、対応するfaiファイル(インデックスファイル)を同じディレクトリに置いておきます。
$ samtools --version samtools 1.3 Using htslib 1.3 # for genome $ samtools faidx genome.fa
マッピングデータの読み込み
メニューバーのFile->Load From File...から、マッピングツールの結果ファイルをロードします。IGVでは様々なフォーマットをサポートしているようですが、今回はBAMファイルをロードしました。(tophat2を使ったマッピング -RNA-seq-)
tophat2のマッピング結果は、accepted_hits.bamとして得られます。IGVで表示しやすいように、samtoolsで事前にsort・indexの付与を済まして置くと良いです。IGVでロードする際、accepted_hits.sort.bamを指定しますが、対応するbaiファイル(インデックスファイル)を同じディレクトリに置いておきます。
$ samtools --version samtools 1.3 Using htslib 1.3 # for mapping data $ samtools sort accepted_hits.bam -o accepted_hits.sort.bam -@ 4 $ samtools index accepted_hits.sort.bam
アノテーションファイルの読み込み
メニューバーのFile->Load From File...から、アノテーションファイルをロードします(IGV:Recommended File Formats)。アノテーションファイルのロードが完了すると、トラックの一番下に遺伝子の位置情報がマッピング情報と一緒に表示されます。
表示記号の意味
IGVの表示は拡大・縮小ができます。様々な情報を分り易く表示するために、ハイライト表示が工夫されています。塩基配列の情報などが確認出来るほど十分に拡大すれば、変異や挿入・欠失などの情報を確認することができます。(IGV:Viewing Alignments)
- 変異リファレンスに対して異なる塩基配列をもつリードの該当塩基は、色分けされてハイライト表示されます。ズームアウトした場合は、縞模様のように表示されます。
- Insertions挿入位置は「I」マークで表示されます。マウスを近くに持っていけば、挿入された塩基情報が表示されます。
- Deletions欠失の位置は、「−」で表示されます。リファレンス配列を参照することで何が欠失しているか判ります。
表示色の意味
マッピング表示エリアの右クリックで、「Color alignment by」を選ぶと、リードのマッピング状況に従って色分けを行うことができます。リードのマークの大半は灰色で表示されますが、緑や赤・青などで色分け表示されるリードに特に注目すると色々な情報が得られるかもしれません。

insert size
ペアエンド間の距離情報にしたがって色分けします。(IGV:Interpreting Color by Insert Size)
- deletion(赤)
- insertions(青)
- ペアとなるリードが他の染色体にマッピングされた場合(様々な色で表現)
pair orientation
ペアエンド間の特徴的な位置や方向にしたがって色分けします。(IGV:Interpreting Color by Pair Orientation)
- Inversions, Inverted Duplication(薄い青・濃い青)
- Tandem Duplication, Translocation on the Same Chromosome(緑)
移動する
IGVの移動は、メニューバーの下のコンボボックスによる染色体の選択・その隣の検索ボックスでの移動が主な移動手段です。特定の場所が表示されたら、マウスでドラッグすることによって近くを自由に移動することが出来ます。
検索ボックス
リファレンスをIGVのプリセット情報を利用したり、アノテーション情報を工夫することで、検索ボックスに遺伝子名などを入力して移動することができます。アノテーション情報などが不足する場合は、場所を直接入力することで特定の場所へ移動することが出来ます。染色体番号:start位置のように入力して「Go」を押します。
ナビゲーション機能
メニューバーの「Regions」を活用すると、移動が便利になります。「Regions」->「Region Navigator」に特定の位置を登録することが出来ます。登録したデータを選択して、「View」ボタンを押すことで特定の場所へ移動が可能です。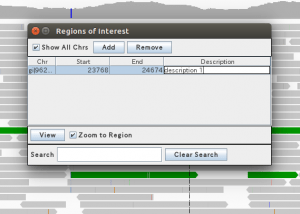
位置をインポートする
たくさん位置を登録したい場合は、データをインポートする方が良いと思います。4つの項目をタブ区切りで記載し、名前.bedとしてファイルを保存します。「Regions」->「Import Regions…」でファイルを指定することで、「Region Navigator」に登録されます。
- 染色体番号
- start
- end
- description
データ例
gi|9626243|ref|NC_001416.1| 0 10 description 1 gi|9626243|ref|NC_001416.1| 0 100 description 2 gi|9626243|ref|NC_001416.1| 100 1000 description 3
こんなときどうする?
- ペアエンドのリードの場所を調べるには?
マッピング表示エリアで、ペアの場所を見たいリードを「コントロールキー」を押しながらクリックします。ペアのリードも同じ色で選択されます。ペアの関係がわかりにくい場合は、右クリックで「View as pairs」を選ぶとより見やすくなる場合があります。

- 見たい場所をサクサク移動したいんだけど?
IGVのメニュー「Regions」->「Region Navigator」に登録するか、「Regions」->「Import Regions…」でたくさんの登録位置を一括で登録します。どちらも、「Region Navigator」で選択して、何度でも簡単に特定の場所に移動することが出来るようになります。上記、「IGVの使い方」の「移動する」を参考にしてください。
- マッピング表示エリアでポップアップを消したいんだけど?
正式なやり方かは分かりませんが、「コントロールボタン」を押している間は、ポップアップが出ません。情報を見たいリードの上でコントロールボタンを離せば情報を見ることができます。
論文・データ解析の支援
個別に直接相談できるのが良いと、たくさんの方にご利用頂いています。


関連記事1


関連記事2














































![バイオインフォ 道場 [bioinfo-Dojo]](https://bioinfo-dojo.net/wp-content/uploads/2016/03/some_object_luca-bravo-alS7ewQ41M8-unsplash.jpg)
